(通讯员 付亮亮 李京津)近日,赵书红教授团队在《BMC Biology》期刊在线发表了题为“Enhancer-promoter interaction maps provide insights into skeletal muscle-related traits in pig genome”的研究成果。该研究在猪上首次同时构建了高分辨率RNA-染色质互作图谱和染色质-染色质互作图谱,并整合近年来已发表的高质量猪产肉相关性状的GWAS结果,鉴定出一批调控猪产肉性状的关键遗传变异及基因,为猪产肉性状遗传机理解析及高效改良提供了支撑。
猪肉占我国居民肉类消费的60%,提高产肉效率是当前猪育种的主要目标。在过去的十多年中,全基因组关联研究(Genome-Wide Association Studies,GWAS)鉴定出了多个与猪产肉性状相关的QTL区域,但是如何进一步挖掘主效基因和因果变异,一直是一项重大挑战。一般来说,基因表达程序与顺式调控元件和染色质相关RNA(chromatin-associated RNA,caRNA)的相互作用密切相关。然而,由于缺乏染色质构象信息(或构象分辨率过低),以及缺少针对caRNA的研究手段,对这些遗传变异进行功能解释成为了猪功能基因组研究的瓶颈问题。
为了解决上述问题,本研究应用GRID-seq技术和课题组自主开发的BL-HiChIP技术,分别构建了高分辨率RNA-染色质互作图谱和染色质-染色质互作图谱,通过联合分析,在全基因组水平解析了caRNA及染色质三维构象在基因表达中的精细调控作用,揭示了使用高分辨率染色质互作图谱探究顺式调控元件与靶基因之间精确调控关系的重要性。同时,运用整合组学策略,对猪15个产肉相关性状(包括饲料转化率、平均日增重、滴水损失等)的GWAS信号进行了解析,鉴定出54个重要候选主效基因和223个突变。该结果为猪产肉性状遗传机理解析奠定了重要基础,也为猪产肉性状改良提供了重要支撑。同时,本研究策略也为猪复杂经济性状遗传机理的解析提供了新思路。
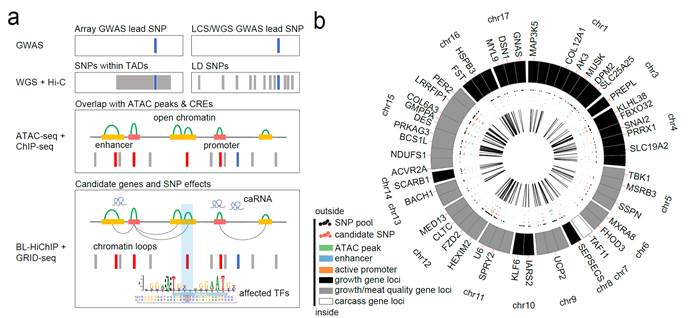
整合组学策略鉴定主效基因及功能突变模式图
本研究是我校赵书红教授团队在猪整合组学知识库ISwine创建及猪全基因组调控元件精细注释研究工作后,利用整合组学策略高效解析猪产肉性状遗传机理的又一重要进展。
我校博士生李京津、项月和张露为论文共同第一作者。我校赵书红教授、李新云教授和付亮亮老师为文章共同通讯作者,上述研究工作得到了国家重点研发计划和国家自然科学基金等项目的资助。
审核人:赵书红
原文链接:https://bmcbiol.biomedcentral.com/articles/10.1186/s12915-022-01322-2

